
细胞周期蛋白依赖激酶靶向库

Cyclin-dependent Kinase Focused Library
产品编号LF1035
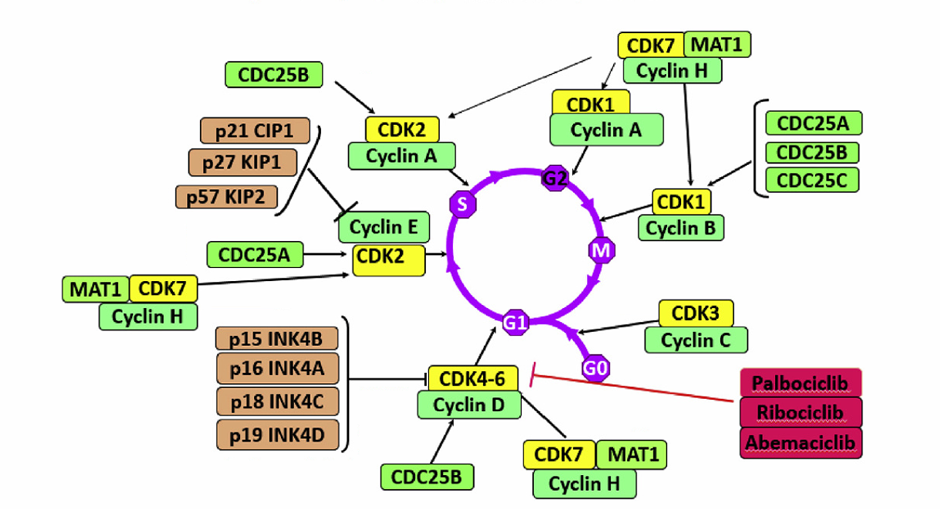
细胞分裂周期由细胞周期蛋白依赖性激酶(CDK)调控,该激酶由催化亚基(cdk1-cdk8)和调节亚基(cyclin A–cyclin H)构成。CDK在细胞周期的G1期到S期检查点中起关键作用(图1)。抑制细胞周期蛋白依赖性激酶是当前抗肿瘤药物研发领域的重要课题。过去十年间,研究人员已发现多类针对该蛋白激酶的小分子抑制剂,并通过X射线晶体学解析阐明了其抑制机制的结构基础。目前已报道的CDK抑制剂包括:黄酮吡醇、苯并咪唑嘧啶类、丁内酯嘌呤衍生物、星形孢菌素、靛玉红、泡诺类化合物等。
我们采用独特策略,设计了靶向CDK的专用筛选库,包含1,700多个类药性筛选化合物:
• CDK7筛选集合
• CDK2靶向集合
我们的内部受体虚拟筛选方法融合了多重技术:类药性过滤、分子对接、重打分、关键分子间氢键与疏水相互作用检测,最终通过配体-受体复合物的可视化验证完成精准筛选。
产品编号LF1035
细胞周期蛋白依赖激酶靶向库规格
- 30 μL x 10 mM (in DMSO)
- 50 μL x 10 mM (in DMSO)
- 100 μL x 10 mM (in DMSO)
- 250 μL x 10 mM (in DMSO)
 产品描述
产品描述
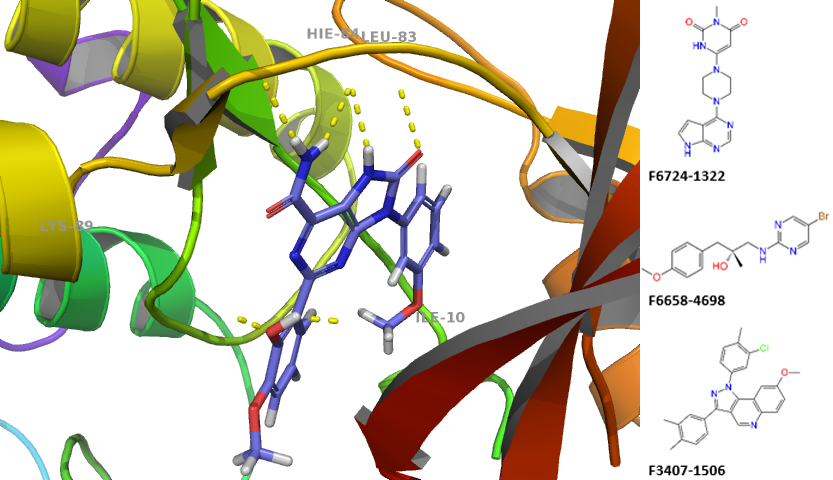
细胞周期蛋白依赖性激酶7(CDK7)筛选库
基于蛋白质数据库收录的CDK7激酶与抑制剂ICCE0943及ATP-γ-S复合物晶体结构,开发了该靶向CDK7的聚焦化合物库。通过基于分子对接的虚拟筛选方案(采用薛定谔Glide软件的SP模式),对内部数据库中潜在的CDK7活性位点结合剂进行系统性筛选。该筛选未设置对接约束条件,以确保SP对接算法能够探索尽可能多的配体构象空间。
此外,本研究联合应用二维指纹相似性搜索技术,以85%的Tanimoto相似度为阈值,对ChEMBL数据库中已知具有CDK7抑制活性的化合物参考集进行筛选。参考集化合物的遴选标准为已报道的最大活性值(IC50/Ki50 < 10 mM)。通过虚拟筛选与相似性过滤的联合策略,最终从1300个类药性筛选化合物中优选出本CDK7激酶筛选库的最终成员。
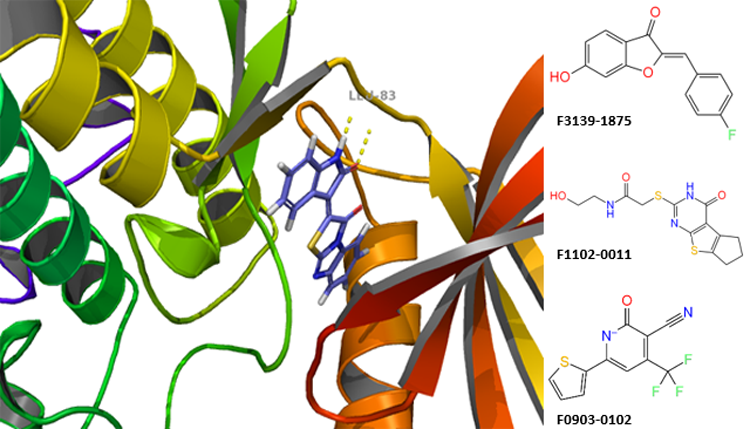
CDK2(细胞周期蛋白依赖性激酶2)靶向化合物库构建方案
初步筛选基于Lipinski规则和Veber规则对内部数据库进行过滤。随后采用分子对接技术,并在DOCK程序中分析对接结果。蛋白质结构的分子动力学模拟与预处理通过GROMACS软件完成,配体分子则使用GAMESS和GROMACS进行处理。最终筛选出400余种小分子化合物,纳入Life Chemicals CDK2激酶筛选库。
 包装和储存
包装和储存
- 可选用DMSO耐受的96/384孔板或2D 条形码编码管;
- 干粉蓝冰运输,DMSO溶液干冰运输;
- 排布:96孔板:1st & 12th 空白对照,384孔板:1st & 2nd & 23th & 24th空白对照。
 包装和储存
包装和储存
- 可选用DMSO耐受的96/384孔板或2D 条形码编码管;
- 干粉蓝冰运输,DMSO溶液干冰运输;
- 排布:96孔板:1st & 12th 空白对照,384孔板:1st & 2nd & 23th & 24th空白对照。
 化合物库定制
化合物库定制

 很棒
很棒