
抗新冠病毒化合物库
Coronavirus Library
产品编号CB1200
严重急性呼吸综合征冠状病毒2(SARS-CoV-2,曾称2019新型冠状病毒2019-nCoV)在全球范围内迅速传播,造成了前所未有的公共卫生负担。开发能够靶向病毒生命周期多个环节的药物,可能成为应对当前及未来冠状病毒大流行的有效策略。
冠状病毒化合物库收录了约15,000种可能与SARS-CoV-2病毒靶点或宿主靶点ACE2相互作用的化合物。该化合物库由哈佛大学瓦格纳实验室合作筛选建立,采用其自主研发并发表的"虚拟流"筛选方法。关于"虚拟流"方法学的详细说明已发表于《自然》期刊(Gorgulla, C., Boeszoermenyi, A., Wang, Z.等."一个开源药物发现平台实现超大规模虚拟筛选"《自然》580卷663-668页,2020年)。这个针对SARS-CoV-2/COVID-19的小分子化合物库也可应用于其他冠状病毒研究项目。
产品编号CB1200
抗新冠病毒化合物库规格
- 1 mg
- 100 μL x 10 mM (in DMSO)
- 250 μL x 10 mM (in DMSO)
 产品描述
产品描述
亮点
• 高质量、无PAINS特征、类先导化合物及类药性小分子化合物
• 适用于SARS-CoV-2/COVID-19及其他冠状病毒研究
• 提供超过15,000种化合物,覆盖17个靶点
方法学
ChemBridge公司库存的130余万种化合物通过Virtual Flow筛选平台,针对17种潜在病毒及宿主靶标的40个不同作用位点分别进行了分子对接。经初步筛选后,进一步剔除不符合要求的化合物,确保冠状病毒化合物库仅包含高质量、类先导或类药性化合物,且不含不良化学基团及PAINS结构警示特征。每个靶标筛选的化合物数量介于1,000至5,000种之间,实际每个位点筛选1,000至2,000种化合物。
ChemBridge库存化合物的结构预处理采用VirtualFlow配体预处理模块(VFLP)完成。每次虚拟筛选均使用单一靶标结构,且蛋白质保持刚性构象。针对刺突蛋白HR1结构域、核蛋白RNA结合界面、nsp12的RNA结合位点以及nsp7和ORF7a等位点,采用QuickVina-W2程序进行盲对接;其余位点特异性对接则使用QuickVina 2程序。两种对接软件均基于AutoDock Vina算法。受体结构通过AutoDockTools工具从PDB格式转换为PDBQT格式。
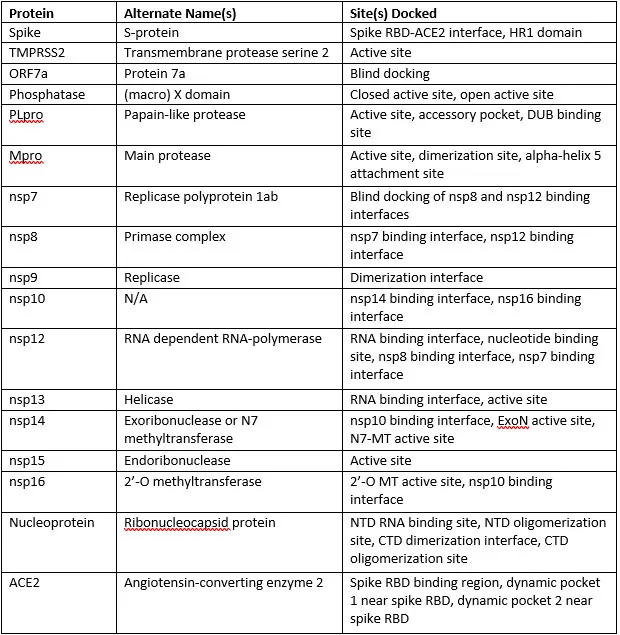
靶点
 包装和储存
包装和储存
- 可选用DMSO耐受的96/384孔板或2D 条形码编码管;
- 干粉蓝冰运输,DMSO溶液干冰运输;
- 排布:96孔板:1st & 12th 空白对照,384孔板:1st & 2nd & 23th & 24th空白对照。
 包装和储存
包装和储存
- 可选用DMSO耐受的96/384孔板或2D 条形码编码管;
- 干粉蓝冰运输,DMSO溶液干冰运输;
- 排布:96孔板:1st & 12th 空白对照,384孔板:1st & 2nd & 23th & 24th空白对照。
 化合物库定制
化合物库定制

 很棒
很棒